Diese Studie stellt die Multidimensionale Antivirale Antikörperdatenbank (MAAD) vor, eine umfassende und standardisierte Plattform, die Sequenz-, Struktur- und Funktionsdaten von Antikörpern gegen drei hochwirksame RNA-Virusfamilien integriert. MAAD dient nicht nur als kuratiertes Datenrepository, sondern auch als interaktives Analysewerkzeug zur Unterstützung des rationalen Antikörper-Engineerings, des strukturorientierten Impfstoffdesigns und der KI-gestützten Antikörperentwicklung.
Wichtigste Punkte:
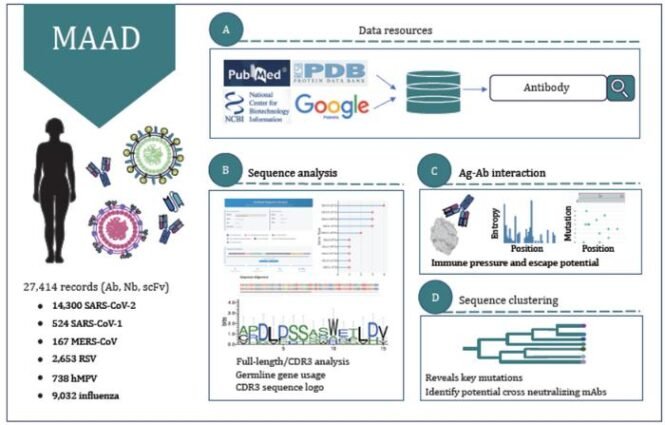
Datenbankinhalt & Umfang: MAAD erfasst und annotiert systematisch 27.414 Einträge zu Antikörpern, Nanobodies und Einzelketten-Variablenfragmenten (scFvs) aus 805 Fachpublikationen und 140 Patenten. Der Fokus liegt auf wichtigen Krankheitserregern aus drei großen RNA-Virusfamilien: Coronaviridae (SARS-CoV-1, SARS-CoV-2, MERS-CoV), Orthomyxoviridae (Influenza) und Pneumoviridae (RSV, hMPV).
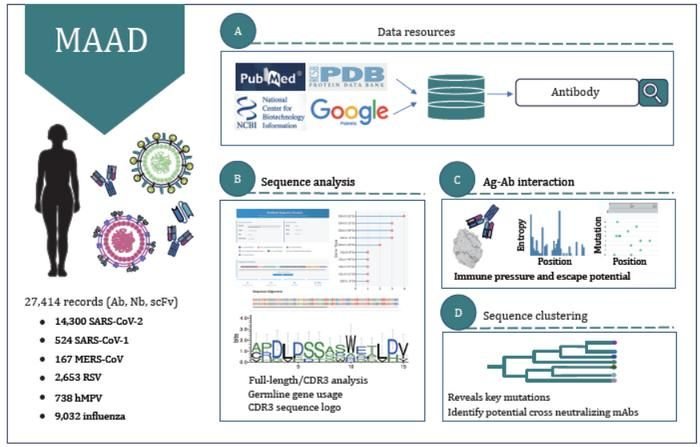
Standardisierte Annotation und Integration: Jeder Eintrag wird mit standardisierten Metadaten angereichert, darunter Sequenzen, Antigenziele, Bindungs-/Neutralisationsprofile, Annotationen der komplementaritätsbestimmenden Region (CDR), Verwendung von V/J-Keimbahngenen, Daten zur somatischen Hypermutation (SHM) und entsprechende Strukturinformationen, wodurch eine echte Integration von Sequenz-Struktur-Funktionsdaten erreicht wird.
Interaktive Analysemodule: Die Plattform ist mit einer Reihe leistungsstarker interaktiver Tools ausgestattet:
Sequenzanalyse: Unterstützt Ähnlichkeitssuchen in voller Länge oder auf CDR-Ebene, die Erstellung von Keimbahn-Gennutzungsprofilen und die Generierung von CDR3-Sequenzlogos.
Strukturanalyse: Integriert 1.394 Antigen-Antikörper-Komplexstrukturen. Ein interaktiver Viewer ermöglicht die Untersuchung von Grenzflächenresten, die mit ortsspezifischer Shannon-Entropie und Mutationshäufigkeit annotiert sind, um Hotspots der Immunflucht zu identifizieren.
Phylogenetisches Clustering: Ermöglicht die Erstellung phylogenetischer Bäume für vom Benutzer bereitgestellte Sequenzen und die Untersuchung vorab berechneter Bäume, wodurch funktionelle Rückschlüsse für nicht validierte Antikörper auf der Grundlage der evolutionären Nähe zu charakterisierten Antikörpern erleichtert werden.
Einzigartige Erkenntnisse und Ergebnisse: Die Analyse deckt sowohl konservierte als auch pathogenspezifische Keimbahngen-Präferenzen auf. Beispielsweise ist IGHV3-21 in RSV-Antikörpern angereichert, während IGHV1-69 und IGHV3-30 von verschiedenen Viren häufig genutzt werden. Die Entropieanalyse von Grenzflächenresten visualisiert Schlüsselregionen viraler Variabilität und potenzieller Immunflucht.
Anwendungen und Bedeutung: MAAD geht über die Rolle einer statischen Datenbank hinaus und dient als dynamische, multifunktionale Plattform für das rationale Antikörper-/Impfstoffdesign, das Training von KI-Modellen und Studien zur Antikörperentwicklung. Die modulare Architektur und das standardisierte Datenschema sind für die zukünftige Integration quantitativer experimenteller Daten (z. B. Bindungsaffinität, DMS-Ergebnisse) und die Erweiterung auf weitere Krankheitserreger ausgelegt.
Gesamtbedeutung:
Durch die Erstellung dieser umfassenden, frei zugänglichen Datenbank mit leistungsstarken Analysefunktionen trägt die Studie wesentlich zum systematischen Verständnis der Sequenz-Struktur-Funktions-Beziehungen antiviraler Antikörper bei. MAAD stellt eine unverzichtbare Datenressource und ein Forschungsinstrumentarium dar, um die Abwehr aktueller und zukünftiger viraler Bedrohungen zu verbessern.

